Il punto sul sequenziamento del virus SARS-CoV-2 con la prof.ssa Mencacci e la prof.ssa Spaccapelo
L’Umbria è autonoma sul fronte del sequenziamento dei tamponi positivi per il virus SARS-CoV-2 e poter individuare in tempo reale eventuali varianti.
Un risultato importante, quello raggiunto dalla Professoressa Antonella Mencacci, direttore della Microbiologica dell’Azienda Ospedaliera di Perugia e professore ordinario di Microbiologia dell’Università di Perugia, dalla Professoressa Roberta Spaccapelo, professore associato presso la stessa Università, e dal loro team.
I campioni non vengono più inviati all’Istituto Zooprofilattico di Teramo per essere sequenziati dato che negli ultimi sequenziamenti fatti in entrambi i laboratori i risultati ottenuti sono risultati perfettamente identici.
Il laboratorio di Microbiologia ha accettato e vinto questa nuova sfida grazie agli studi e all’expertise maturati in questi mesi da parte del personale e ai tecnici appositamente formati, riuscendo a procedere autonomamente e aprendo la strada a nuove possibilità sul fronte della diagnostica microbiologica.
“Si potranno sequenziare non solo i virus, ma anche casi particolarmente gravi di infezioni, casi dovuti a batteri multiresistenti, o casi rari di cui non si conosce l’agente eziologico”, spiega la prof.ssa Mencacci.
Ma come funziona il sequenziamento?
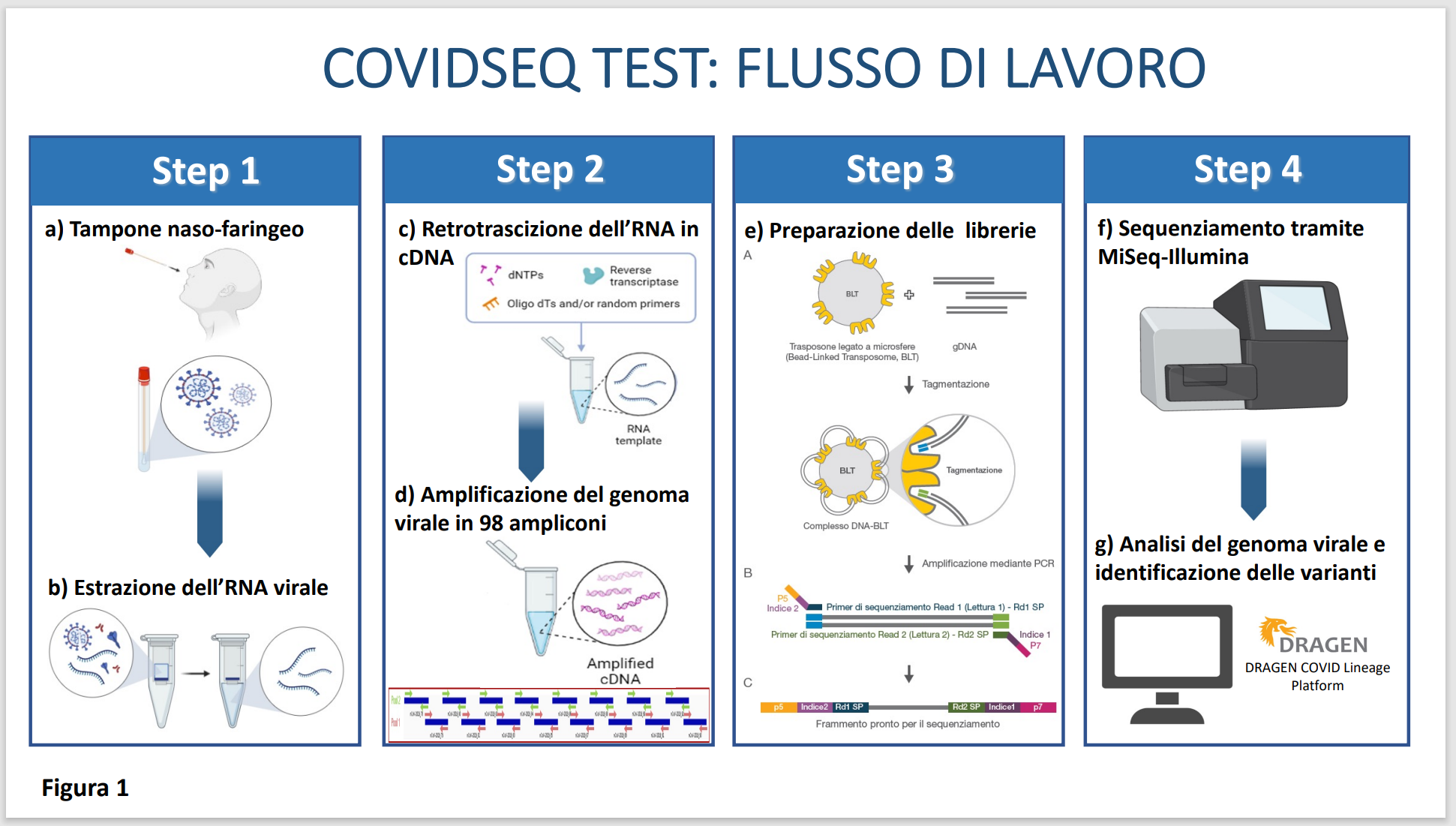
“Il primo step è la selezione di quei campioni positivi al virus che meritano di essere sequenziati (ad esempio quelli che hanno dato luogo ad un caso clinico molto grave). Alcuni campioni vengono invece scelti casualmente, per individuare la eventuale comparsa di varianti non ancora note. Una volta selezionati, bisogna estrarne l’RNA virale, cioè il genoma del virus” (Figura 1).
Il processo di retrotrascrizione.
Il secondo step prevede la retrotrascrizione dell’RNA virale in DNA complementare (cDNA), in quanto l’RNA non può essere direttamente sequenziato.
Preparazione delle librerie genomiche e sequenziamento dei campioni.
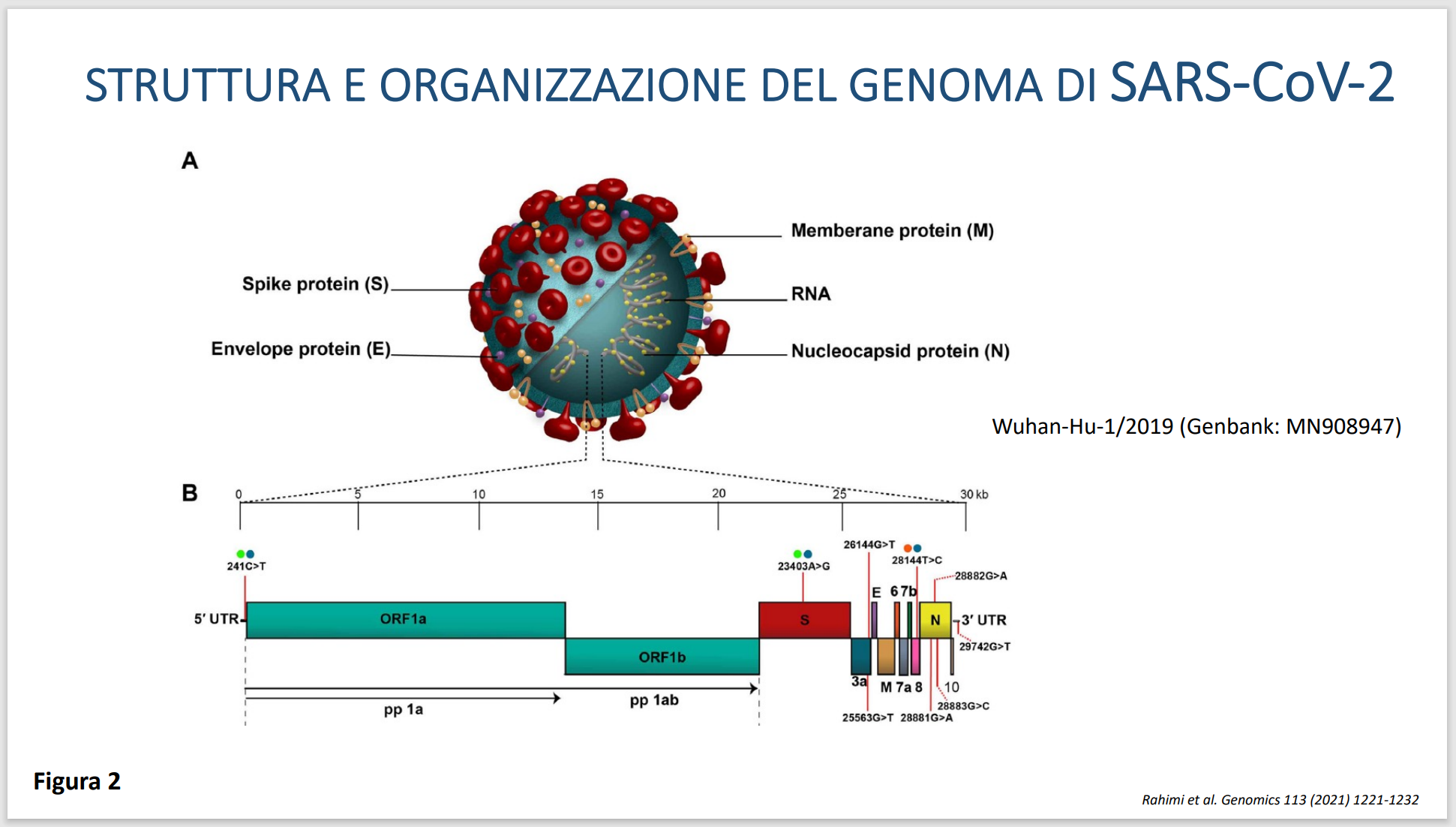
A spiegare gli step successivi è la prof.ssa Spaccapelo: “Il genoma del virus SARS-CoV-2 è molto piccolo rispetto al genoma di una cellula umana, consta di circa 30mila paia di basi (Figura 2), ma per essere sequenziato è necessario amplificarlo in tanti piccoli frammenti di circa 300 paia di basi ciascuno (98 ampliconi in totale). Il terzo step prevede la preparazione della libreria (Figura 1).
Nel quarto step le librerie preparate sono sequenziate con la piattaforma MiSeq (Illumina) che si trova al CREO.
Il risultato del sequenziamento – continua la prof.ssa Spaccapelo - viene automaticamente caricato in una piattaforma online all’interno della quale abbiamo a disposizione il software DRAGENTM COVID Lineage che ci permette di riallineare i 98 frammenti, ricreare la sequenza completa del genoma SARS-CoV-2 per poi confrontarla con quella del ceppo originario identificato a Wuhan (Cina). Grazie a questo software possiamo identificare le varianti e generare una sequenza consenso del genoma (Figura 3)
Allineamento sequenze del gene Spike (S) per l’identificazione delle varianti

I tamponi che il Laboratorio di Microbiologia riceve e poi seleziona arrivano dagli ospedali e dai distretti dell’Umbria. Un team composto dalla Dr.ssa Barbara Camilloni e 4 tecnici si occupa delle varie fasi del processo, dalla selezione dei campioni da sequenziare, all’estrazione del materiale al sequenziamento, fino all’analisi dei dati e al caricamento sulle piattaforme informatizzate. Variano dai 48 ai 56 i campioni sequenziati a settimana e il processo dura 4 giorni, di cui 30 ore per lo strumento dedicato.
“Sequenziamo casi particolarmente gravi, le reinfezioni in breve tempo dall’infezione già risolta, le infezioni dopo la terza dose di vaccino oppure cluster o campioni che mostrano qualche curva diversa al test molecolare- conclude la prof.ssa Mencacci – ma il sequenziamento è importante anche per studiare altri virus e batteri, non solo per SARS-CoV-2. Per questo voglio ringraziare la Regione Umbria, l’Azienda Ospedaliera e l’Università degli Studi di Perugia, per il supporto e la fiducia che ci hanno dato”.